È di Melody Di Bona, PhD UniGe, uno dei migliori lavori della Biophysical Society
Melody Di Bona, con la Laurea Magistrale in Biotecnologie medico-farmaceutiche (110 e lode con dignità di stampa) e il Dottorato in Fisica conseguiti all'Università di Genova, è ora postdoc al Memorial Sloan Kettering Cancer Center di New York (USA).
Un paper della nostra PhD, primo autore insieme a Michael A. Mancini, Davide Mazza, Giuseppe Vicidomini, Alberto Diaspro e Luca Lanzanò, è stato inserito tra i migliori lavori della Biophysical Society, riportato nel volume Biophysical Journal – best of 2019. L’articolo si intitola "Measuring Mobility in Chromatin by Intensity-Sorted FCS", in italiano "Misurazione della mobilità nella cromatina mediante FCS a intensità differenziata".
La dott.ssa Di Bona ci spiega in cosa consiste la sua ricerca e quali risultati sono oggetto dell’articolo annoverato tra i migliori lavori del 2019 in ambito biofisico.

Cromatina : cos'è e qual è la sua funzione?
Il nucleo delle cellule eucariotiche contiene il DNA, ovvero il patrimonio genetico dell’individuo, organizzato in una struttura condensata definita cromatina.
Recentemente è stata evidenziata una correlazione tra il grado di compattezza della cromatina, e dunque la sua organizzazione strutturale, e la sua funzione nell'esprimere correttamente l’informazione contenuta nel DNA. Nonostante ciò, essendo il nucleo un ambiente estremamente denso e compatto, risulta difficile da studiare con metodi convenzionali.
Come si studia la cromatina?
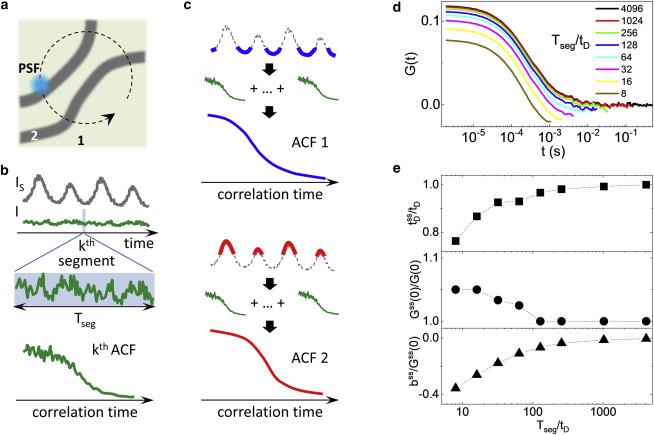
Un modo indiretto per investigare quanto e come la cromatina è compattata è usare la Spettroscopia a Correlazione di Fluorescenza (FCS), una tecnica che studia la fluttuazione, in un volume di osservazione microscopico (femtolitri, pari a 10⁻¹⁵ litri), della fluorescenza. Tale fluttuazione è dovuta all'ingresso e all'uscita nel volume di osservazione di proteine fluorescenti, ed è tipica di ogni proteina: l’analisi matematica della correlazione tra le fluttuazioni permette di ricavare la velocità a cui la proteina fluorescente si muove (coefficiente di diffusione). Tramite il coefficiente di diffusione si può capire se un ambiente è più compatto (la proteina si muove più lentamente), o se viceversa è meno denso.
Tuttavia, uno dei maggiori problemi riscontrati con questa tecnica è la mancanza di informazione spaziale, che è invece essenziale nello studio dell’organizzazione del nucleo, ma anche la mobilità intrinseca delle cellule e delle strutture cromatiniche, che interferisce con una corretta misurazione spazio-temporale del coefficiente di diffusione.
Un nuovo metodo d’indagine
Nell'articolo pubblicato su Biophysical Journal, il gruppo di ricerca della dott.ssa Di Bona ha presentato una tecnica innovativa che utilizza FCS muovendo il volume di osservazione all'interno del nucleo e sfruttando un secondo canale di acquisizione che rappresenta un marker spaziale (Intensity-Sorted FCS). In questo modo è possibile correlare la fluttuazione della fluorescenza con il marker spaziale in ogni momento della misura. Come marker viene utilizzata una molecola fluorescente che lega il DNA, in modo da sapere con esattezza dove la cromatina è più compatta (elevata intensità del marker) o più lassa, e poter quindi ricavare il coefficiente di diffusione di una proteina inerte in quel preciso ambiente nucleare.
Importanti risultati
Per la prima volta i ricercatori sono riusciti a misurare con esattezza la differenza di velocità di una piccola proteina in diversi ambienti nucleari e a tracciare queste dinamiche in modo preciso e accurato anche a seguito di trattamenti che influenzano la compattezza del contenuto nucleare. Inoltre questa tecnica originale è stata applicata allo studio della mobilità di un fattore di trascrizione nucleare in prossimità del suo sito di legame, distinguendo le proteine legate da quelle in movimento. Infine è stata dimostrata la compatibilità di Intensity-Sorted FCS con la tecnica di super-risoluzione SPLIT-STED che permette di osservare volumi ancora più piccoli e di seguire la variazione del coefficiente di diffusione a seconda della variazione del volume di osservazione, fornendo informazioni riguardo alla presenza o meno di ostacoli alla diffusione delle proteine.
Una svolta nello sviluppo di farmaci antitumorali
Il laboratorio della dott.ssa Di Bona si occupa di studiare un particolare gruppo di tumori molto diffuso, caratterizzati da instabilità cromosomica e dalla presenza nel citoplasma di micronuclei, che, dopo rottura, espongono a enzimi citoplasmatici il DNA contenuto al loro interno. Tra questi enzimi, uno in particolare dà inizio ad un pathway tipico dell’immunità innata, cGAS-STING pathway, che se attivato in modo cronico in cellule tumorali porta a metastatizzazione e malignità.
In particolare, la ricerca si concentra sul capire perché i micronuclei si rompano; questo è il passaggio fondamentale per l’attivazione del pathway.
Oltre a metodi di biologia cellulare e molecolare, si utilizzano tecniche di microscopia a super risoluzione e soprattutto FCS per chiarire il ruolo del trasporto citoplasma-micronucleo nel collasso della membrana micronucleare.
Intensity-Sorted FCS può fornire informazioni su come particolari proteine si muovano attraverso il Nuclear Pore Complex.
Una volta capito come il pathway è attivato, si potranno aprire interessanti porte per lo sviluppo di nuovi farmaci antitumorali atti a bloccare l’insorgere di metastasi in questi tipi di tumore.
L’articolo "Measuring Mobility in Chromatin by Intensity-Sorted FCS" è liberamente consultabile al link https://www.cell.com/biophysj/fulltext/S0006-3495(19)30130-4 (DOI:https://doi.org/10.1016/j.bpj.2019.02.003)